此内容仅用于个人学习和研究
重点、重点、重点(第一次用请看3遍以上):
TCGA改版之后,TCGA下载转录组数据页面只有STRA-Counts,处理方法也更新了。
2022年03月改版前:3个步骤(02.转录组数据下载、03.转录组数据整理、04.id转换)+3个脚本处理(moveFiles.pl、merge.pl、symbol.pl)。
2022年03月改版后:2个步骤(03.转录组数据下载、04.转录组数据整理)+1个新脚本(merge.pl)就可以完成以前的3个步骤了,一步整理好到注释好的矩阵(不需要moveFiles.pl、symbol.pl)。新的一个脚本merge.pl就等同于旧版的三个脚本了。
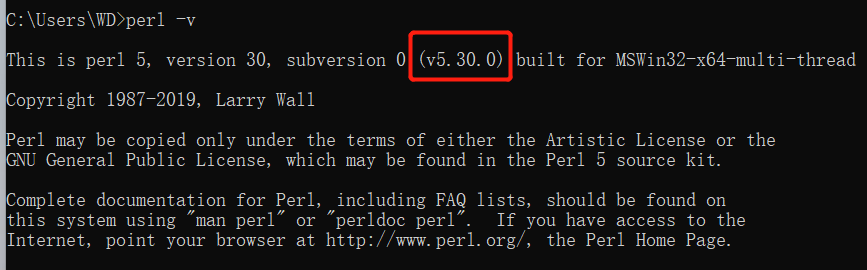
2022年3月29日之后下载的TCGA数据库必须使用新脚本,使用新脚本(merge.pl)之前,必须卸载之前的perl软件,然后重启电脑,然后必须安装草莓版perl 5.30.0(这个我会提供在网盘链接里面的,必须一定只能是perl 5.30.0版本)。
必须卸载之前的perl软件,然后重启电脑,然后必须安装草莓版perl 5.30.0,只能是win系统。
必须卸载之前的perl软件,然后重启电脑,然后必须安装草莓版perl 5.30.0,只能是win系统。
(以下内容作为拓展,不是必须看)TCGA数据库改版下载转录组数据变了只有STAR–Counts
2022年3月29日,TCGA数据库改版了。
TCGA gdc网址:https://portal.gdc.cancer.gov/
选择菜单栏的Repository,进入数据选择页面
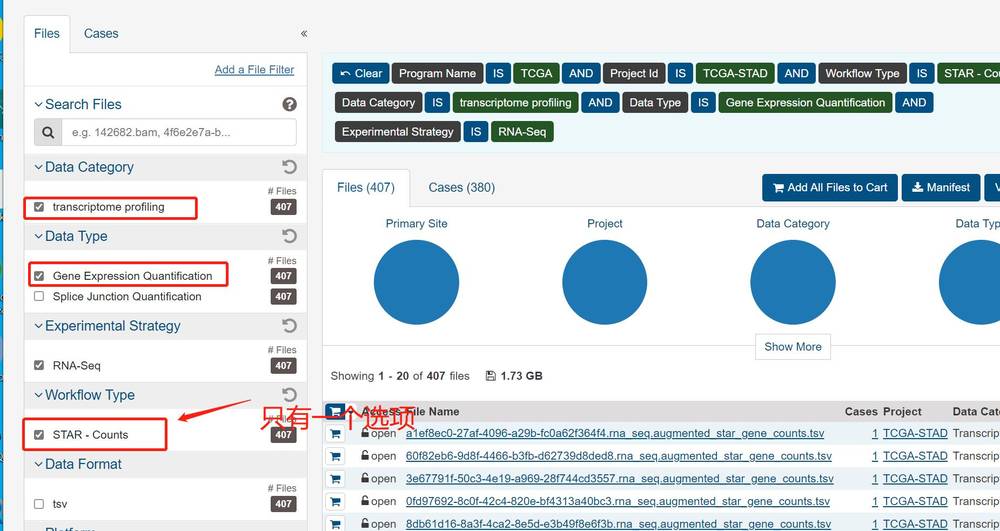
首先选择肿瘤类型,选到Cases,“Program”选择TCGA,“Project”选择自己研究的肿瘤类型,比如这里选择TCGA-STAD(也就是经常用到的胃癌)

接下来选择文件类型Files,“Data Category”选择转录本“transcriptome profiling”,“Data Type”选择Gene Expression Quantification,接下来到了选择“Workfolw Type”,现在只有一个选项,就是STAR – Counts。
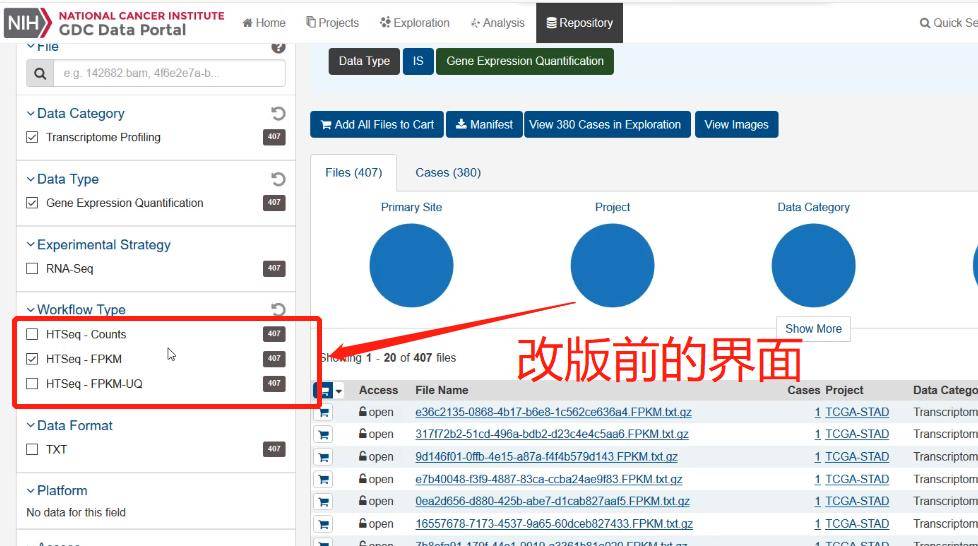
之前的界面是有三个选择的。经过测试,STAR – Counts里面的数据是改变了,所以方法也需要改变,提取symbol.txt的方法相继也需要改变的。
作为一名科研工作者,时时刻刻都在和时间赛跑。吾必须时时刻苦钻研科研,掌握现代科研的知识和技能。愿在未来,不忘初心,牢记使命。只争朝夕,不负韶华,为成为一名优秀的科研工作者而努力,努力践行科学发展观,认真学习科学发展观努力做好本职工作。
科学技术是人类社会发展的原动力。要将“做科研”培养成为一种兴趣,将实践和科研思维结合进行科学创新,才能真正推进科技进步。要如何做研究?--从实践中发现问题;运用知识储备提出问题;依赖先进的技术研究问题;最终的研究成果,要不忘初心,应用于解决实践中的问题上。
读书学习之道,看似无边无际,不知何去何从,倘若勤奋刻苦,书山自有石经,学海岂无彼岸。故不积跬步,无以至千里;不积小流,无以成江海。作为科研工作者,吾应发奋图强,刻苦学习!方能在科研方面更进一步!
此内容为个人学习笔记,外人禁止查看

